Rolle von SIRT7 bei der FLT3-ITD-vermittelten Alterung von hämatopoetischen Stammzellen
Onkogenes FLT3 ITD kontrolliert HSC Differenzierung über Sirt7
Grafik: Jörg P. MüllerForschungsthemen
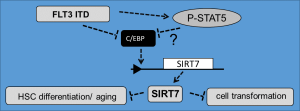
SIRT7 wurde als Faktor identifiziert, der die Quiescense und Differenzierung hämatopoetischer Stammzellen (HSC) reguliert. Eine Suppression von SIRT7 führt zur Initiierung der HSC-Differenzierung. Unsere Studien zeigen ein reduziertes SIRT7-Niveau in Zellen, welche die onkogene Rezeptortyrosinkinase FLT3-ITD exprimieren. Die Unterdrückung von SIRT7 wird hier durch die aberrante FLT3-ITD-Kinaseaktivität kontrolliert. Die Rekonstitution von SIRT7 in FLT3-ITD-Zellen unterdrückte die Zelldifferenzierung, die Geninaktivierung stimulierte sie. AML-Patienten mit niedriger SIRT7-Expression haben eine schlechte Prognose, was die klinische Relevanz dieses Proteins für das Überleben der Patienten zeigt (Kaiser et al., Leukemia 2020Externer Link).
Durch die Verwendung von in-vitro- und in-vivo-Systemen untersuchen wir den Mechanismus, wie FLT3 ITD die HSC-Alterung und -Differenzierung über SIRT7 in leukämischen Zellsystemen steuert. Die in vivo-Rekonstitution von transduzierten BM-Zellen mit veränderten SIRT7-Aktivitäten in FLT3-ITD-Mäusen ermöglicht Einblicke in den Mechanismus, wie SIRT7 die HSC-Alterung und die Entwicklung der myeloproliferativen Erkrankung und der klonalen Expansion beeinflusst. Die Beschreibung der zugrundeliegenden Signalwege, welche die Alterung und Differenzierung von HSC vermitteln, liefert ein weitergehendes Verständnis in die Pathogenese hämatologischer Erkrankungen und wird neue Wege zur Beeinflussung der Leukämieentwicklung eröffnen.
Methoden
In vitro-Techniken
- molekularbiologische Manipulation von Zelllinien
- transiente und Virus-vermittelte stabile Unterdrückung und Überexpression von Genen
- CRISPR/Cas-vermittelte Inaktivierung von Genen (genspezifisch, genomweit)
- CRISPRa-, CRISPRi-Anwendungen
- Bildgebende Verfahren zur räumlichen und zeitlichen Proteinlokalisierung und Proteinkomplexbildung (FRET)
In vivo-Techniken:
- Mausmodelle, die Onkogene überexprimieren
- Zelltransduktion in syngenen Mausmodellen
- Knochenmarktransduktion und -transplantation
- CRE-vermittelte gewebespezifische Inaktivierung von Genen
- pharmakologische Anwendungen an Mäusen
Ausgewählte Publikationen
Bär I, Ast V, Meyer D, König R, Rauner M, Hofbauer LC, Müller JP. (2020) Aberrant Bone Homeostasis in AML Is Associated with Activated Oncogenic FLT3-Dependent Cytokine NetworksExterner Link. Cells, 9(11):2443
Müller JP, Schmidt-Arras D. (2020) Novel Approaches to Target Mutant FLT3 Leukaemia.Externer Link Cancers (Basel), 12(10)
Kaiser A, Schmidt M, Huber O, Frietsch JJ, Scholl S, Heidel FH, Hochhaus A, Müller JP, Ernst T. (2020) SIRT7: an influence factor in healthy aging and the development of age-dependent myeloid stem-cell disorders.Externer Link Leukemia, 34(8):2206-2216
Kellner F, Keil A, Schindler K, Tschongov T, Hünninger K, Loercher H, Rhein P, Böhmer SA, Böhmer FD, Müller JP. (2020) Wild-type FLT3 and FLT3 ITD exhibit similar ligand-induced internalization characteristics.Externer Link J Cell Mol Med, 24(8):4668-4676
Kresinsky A, Schnöder TM, Jacobsen ID, Rauner M, Hofbauer LC, Ast V, König R, Hoffmann B, Svensson CM, Figge MT, Hilger I, Heidel FH, Böhmer FD, Müller JP. (2019) Lack of CD45 in FLT3-ITD mice results in a myeloproliferative phenotype, cortical porosity, and ectopic bone formationExterner Link. Oncogene, 38(24):4773-4787