Molekulare Zytogenetik
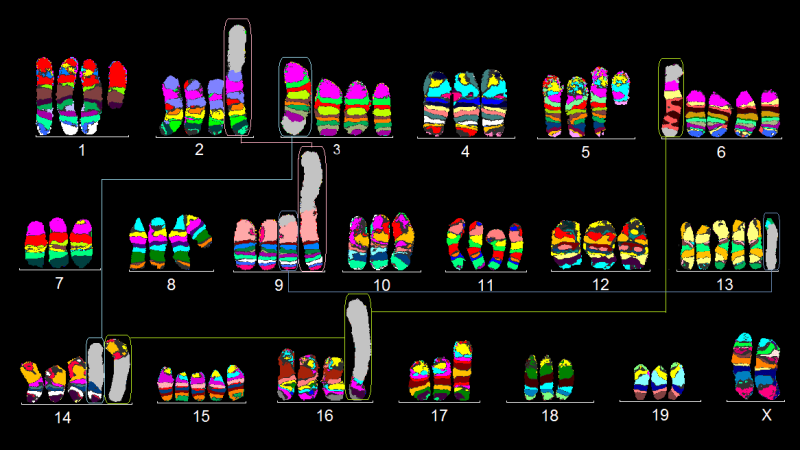
Murine Multicolor Banding (mcb) wurde auf Chromosomen der JC Brustkrebs-Zelllinie angewandt.
Grafik: Thomas Liehr aus Azawi et al. (Mol Cytogenet, 2021)Forschungsthemen
Cytogenomische Charakterisierung von murinen Tumorzelllinien und humanen Leukämien
In der Arbeitsgruppe Molekulare Zytogenetik liegt eine weltweit einmalige Expertise und DNA-Probensammlung vor, die geeignet ist, kryptische genetische / chromosomale Umbauten besser zu erfassen als viele andere Verfahren. Diese Proben (z. T. selbst erstellt mittels Glasnadel-basierender Chromosomenmikrosezierung) werden in, je nach Fragestellung individuellen Probensets zusammengestellt und in Vielfarben-Fluoreszenz-in-situ-Hybridisierung (FISH) eingesetzt. Die murinen Proben ermöglichten in den letzten Jahren die erstmalige cytogenomische Charakterisierung von etwa 20 murinen Tumorzelllinien. Weiterhin wurden und werden in internationalen Kooperationen ansonsten nicht aufklärbare komplexe humane Leukämiefälle oder bestimmte solide Tumormodelle bearbeitet. All das mündete und mündet in einer Vielzahl von PublikationenExterner Link.
Methoden
Die Arbeitsgruppe Molekulare Zytogenetik ist seit > 20 Jahren auf Vielfarben-Fluoreszenz-in-situ-Hybridisierungs (FISH)-Verfahren spezialisiert und eines der wenigen Labore weltweit, die Glasnadel-basierende Chromosomenmikrosezierung durchführen können. In Zusammenarbeit mit dem Leibniz-Institut für Alternsforschung – Fritz-Lipmann-Institut (FLI)Externer Link steht außerdem die Technik des molecular combings zur Verfügung. Bänderungszytogenetik und array-comparative genomic Hybridization (aCGH9) stehen für entsprechende Fragestellungen in Nachbarlaboren (Dr. Weise, Dr. Mrasek) innerhalb des Instituts für HumangenetikExterner Link bereit.
Ausgewählte Publikationen
Capela de Matos RR, Othman M, Ferreira GM, Monteso K, de Souza MT, Rouxinol M, Melo JB, Carreira IM, Abdelhay E, Liehr T, Ribeiro RC, Silva M. (2022) Somatic homozygous loss of SH2B3, and a non-Robertsonian translocation t(15;21)(q25.3;q22.1) with NTRK3 rearrangement, in an adolescent with progenitor B-cell acute lymphoblastic leukemia with the iAMP21.Externer Link Cancer Genet, 262-263:16-22
Koltsova AS. Efimova OA, Malysheva OV, Osinovskaya NS, Liehr T, Al-Rikabi A, Shved NY, Sultanov IY, Chiryaeva OG, Yarmolinskaya MI, Polenov NI, Kunitsa VV, Kakhiani MI, Tral TG, Tolibova GK, Bespalova ON, Kogan IY, Glotov AS, Baranov VS, Pendina AA. (2021) Cytogenomic profile of uterine leiomyoma: In vivo vs. in vitro comparison.Externer Link Biomedicines, 9(12):1777
Kadioglu O, Saeed MEM, Mahmoud N, Azawi S, Mrasek K, Liehr T, Efferth T. (2021) Identification of novel drug resistance mechanisms by genomic and transcriptomic profiling of glioblastoma cells with mutation-activated EGFR.Externer Link Life Sci, 284:119601
Piaszinski K, Rincic M, Liehr T, Azawi S. (2021) Molecular cytogenetic characterization of the murine melanoma cell lines S91 clone M3 and B16-F1 with variant B16-4A5. Externer LinkCytogenet Genome Res, 161(1-2):82-92
Azawi S, Rincic M, Liehr T. (2021) Cytogenomic characteristics of murine breast cancer cell line JC.Externer Link Mol Cytogenet, 14(1):7
Links
Mehr InformationenExterner Link
Homepage von Thomas LiehrExterner Link